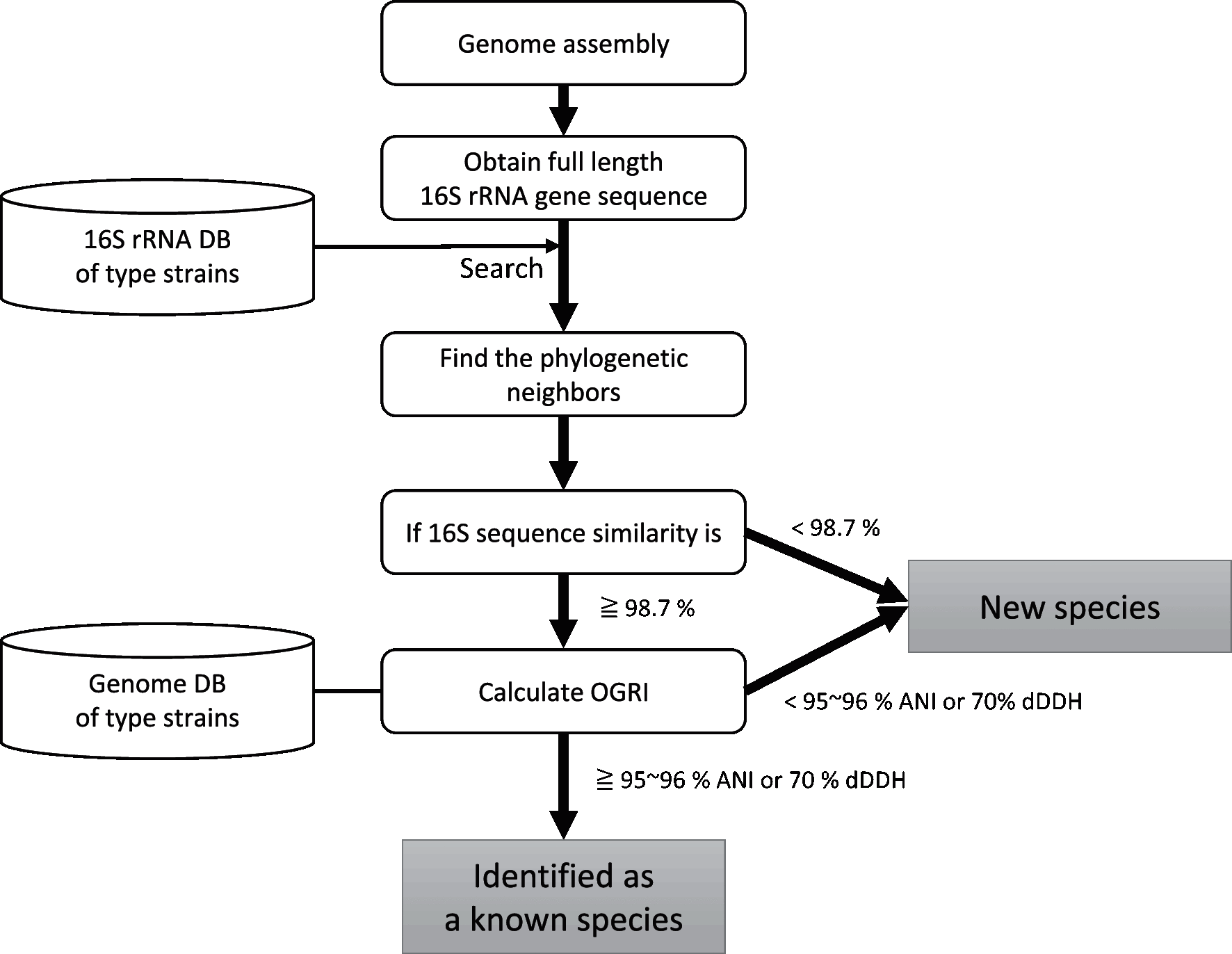
내가 생각하는 진화라는 것은 결국 유전체 서열의 변화가 표현형으로 나타나고, 환경에 가장 잘 어울리는 개체의 빈도가 집단 내에서 늘어나는 현상이다. 좀더 포괄적으로 말하자면 적합도(fitness)가 낮아진 개체의 빈도가 줄어드는 것까지 포함해야 할 것이다.
흔히들 박테리아의 진화라고 하면 단일 염기서열 수준에서 일어나는 염기치환변이 또는 indel이 가장 큰 비중을 차지할 것으로 생각하기 쉽다. 상동유전자 서열을 여러 종에서 모은 다음 다중서열정렬을 해 보면 기준 종에서 먼 것일수록 염기서열의 차이가 더 크게 나타난다. 이러한 염기(서열)의 변화가 일어나는 원인은 무엇일까? DNA polymerase의 실수, radiation으로 손상된 염기의 불완전한 수복, DNA의 double-strand break가 일어난 뒤 일어나는 error-prone repair 등 다양한 메커니즘이 있지만 교과서를 쓰고자 함이 아니니 깊게 들어가지는 않겠다.
실제로는 이보다 과격한(?) 유전체 변이가 더욱 빈번하게 벌어진다. Insertion sequence의 삽입이라든가 prophage의 침입 등이 그러하다. Horizontal gene transfer도 많은 연구자들의 흥미를 끄는 주제이다.
그런데 (homologous) recombination에 의해서 외부의 유전자(일부, 전부 혹은 클러스터)가 들어올 수 있다는 사실을 종종 간과하게 된다. Recombination, 즉 재조합이란 기원이 다른 DNA 조각이 서로 교차하여 새로운 혼성체를 만드는 것을 의미한다. ds DNA 가닥이 끊어져서 서로 근본이 다른 것끼리 연결되는 것이다. 재조합이 일어나는 정도는 미생물 종마다 매우 다르다. 결핵균처럼 매우 제한된 환경에서 '조용히' 서식하는 것에서는 그 정도가 매우 적지만, 자연적인 transformation이 가능한 세균으로서 자유롭게 바글거리며 사는 세균은 그 빈도가 매우 높다.
Recombination은 계통발생학 분석을 하는데 장애가 되기도 한다. 수십 종의 미생물 유전체 서열로부터 multiple sequence alignment를 공들여 얻은 뒤 phylogenetic tree를 그리는 것이 요즘 genomics를 하는 사람들의 공통된 일인데, 유전체 상의 특정 block이 재조합에 의해서 '훌러덩' 유입된 것이라면 족보를 그리는 일이 아주 까다로와지기 때문이다. 그리고 재조합의 검출하는 일 역시 매우 어렵다.
Homologous recombination을 검출하려는 체계적인 노력은 Xavier Didelot의 ClonalFrame이 그 시초가 아닐까 한다(Inference of bacterial microevolution using multilocus sequence data. Genetics 2008, PMC
링크). 이것은 원래 mltilocus sequence 데이터를 이용하는 프로그램이었다.
 |
| 녹색은 mutation, 빨강색은 recombination. |
이후 수백 개의 genome sequence를 다룰 수 있는 도구가 등장하면서 선택의 폭은 더욱 넓어졌고, 요즘 유행인 베이지언 기법을 이용한 것도 등장하게 되었다. 임상에서 분리한 Streptococcus pneumoniae의 대규모 유전체 분석을 통하여 recombination이 매우 빈번하게 일어남을 밝힌 선구적인 논문이 이미 2011년에 Science에 게재된 바 있다.
Rapid pneumococcal evolution in response to clinical interventions. Science 2011 331:430 PubMed
링크
치료 중인 환자에게서 분리된 감염균은 살아남기 위하여 온갖 전략을 구사한다. 그 중의 하나는 항원으로 작용하는 capsule의 스위칭을 하는 것이고, 바로 여기에 recombination이 크게 기여한다는 것이다. 물론 recombination을 검출하고 멋지게 그림을 그려주는 도구는 이 논문 이후에 본격적으로 개발된 것으로 안다. 당시에는 SNP의 density를 이용하여 recombination이 일어난 곳을 추정하였었지만 지금은 훨씬 세련된 통계학적 추론을 이용하는 도구도 있다. 아래 그림의 오른편에 보인 heatmap에서 빨강 블록으로 표시된 것이 바로 재조합이 일어난 곳이다.
요즘 내가 테스트하는 것은 Sanger Institute에서 개발한
Gubbins이다(
링크). 2014년 Nucleic Acids Research에 'Rapid phylogenetic analysis of large samples of recombinant bacterial whole genome sequencing using Gubbins(
링크)'라는 논문으로 발표가 되기도 하였었다. 프로그램 개발을 위한 데이터셋은 바로 위에서 소개한 Science 2011년도 논문의 것을 사용하였다(PMEN1 dataset
링크).
Gubbins를 실행하려면 FASTA 형식의 whole genome alignment가 필요하다. 그런데 이것이 생각처럼 쉽게 뚝딱 만들어지지가 않는다. 먼저 고려할 것은 raw sequencing read에서 시작할 것인가, 혹은 assembled sequence에서 시작할 것인가이다. 처음에는
Roary(assembly 필요)가 만들어내는 core gene alignment를 활용할 생각을 했었으나, 이것은 유전자들을 concatenation한 것이다. 따라서 gubbins 분석이 끝난 뒤 재조합이 일어난 부위의 유전자를 찾으려면 별도의 파일(core_alignment_header.embl)을 참조해야 한다. 이것은 상당히 성가신 일이다. Reference의 좌표에 맞추어서 각 alignment를 배열하고 그 사이에 gap('---')을 넣은 파일이 필요한데 말이다.
Harvest suite의 parsnp를 사용하면 유전체 서열에서 유래한 alignment가 나오지만 이것 역시 concatenated sequence이다. Gubbins가 딱 필요로하는 형태의 FASTA alignment 파일을 만들 방법이 없는지 구글링을 반복하다가
Snippy라는 도구를 찾았다. 이것은 raw sequencing read뿐만 아니라 contig도 사용할 수 있으며, .aln(core SNP alignment)과 .full.aln(whole genome SNP alignment including invariant sites)를 생성해 주므로 후자를 gubbins에 투입하면 된다. 하지만 막상 설치를 하니 에러가 발생하기 시작한다.
첫번째 에러에 대해서는 개발자 사이트에 이미 보고가 되어 있었고(
error with gbk #154) 임시방편으로 버전을 낮추어 쓰라고 하였다. 그런데 이렇게 했더니 vcflib의 위치와 관련한 에러가 또 발생하여 freebayes-parallel 스크립트의 40번째 줄을 고치는 수고까지 해서 겨우 성공적으로 실행을 하였다. Snippy 설치 이력에 대한 정보는 별도의 글로 상세하게 작성해 보고자 한다.