 |
| 왼쪽부터 6LQ8 푸시풀, 6J6 푸시풀(이것은 이영건 선생님 2018년 제작), 그리고 6N2P + 43 싱글. |
이번에는 오랜만에 본업과 관계된 글을 좀 써보려 한다. 인간의 각 장기에 존재한 microbial signature가 다르다는 논문을 읽어보고 이에 대해서 뭔가 글을 쓰려는 강력한 동기에 사로잡혔으나 당장 급한 일 때문에 일단 소개만 해 두고 나중을 노려야 되겠다. Nature의 자매지가 하도 많아서 이제는 다 외지 못할 지경이 되었다.
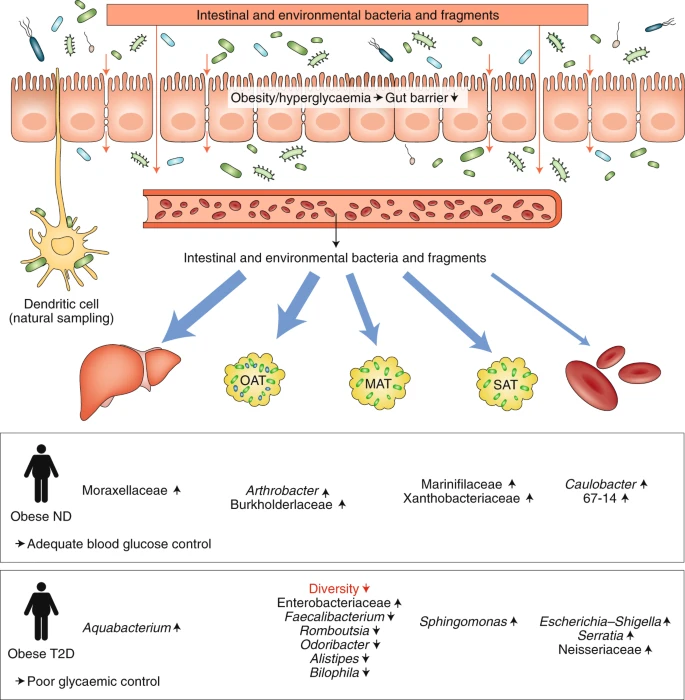
Type 2 diabetes influences bacterial tissue compartmentalisation in human obesity. Nature Metabolism volume 2, pages 233–242(2020) 링크(전문 공개)
Akkermansia muciniphila의 연구로 유명한 Patice D. Cani가 이 저널의 News & Views에서 이 논문을 함축적으로 소개하였으므로 그것을 살펴보는 것도 도움이 된다. 제목은 'Microbial signatures in metabolic tissues: a novel paradigm for obesity and diabetes'(링크)이다. 3월 9일에 게재되었으니 정말 최신 연구이다. 해당 기사의 요약을 그대로 인용한다.
By analysing microbial profiles in three adipose tissue depots and the liver and plasma of morbidly obese individuals, a new study uncovers a unique organ-specific microbial signature, or potential internal ‘tissue microbiota’, in obese people with diabetes.
 |
| 출처: Nature Metabolism |
건강 상태가 나빠지면 gut barrier가 느슨해지면서 장내 미생물이 혈류를 타고 전신을 돌게 된다. Anhê 등은 혈장과 대사 조직에서 이러한 미생물의 '잔해'를 매우 정밀하게 측정하여 서로 다르게 분포한다는 것을 밝혔다. 여기서 잔해라 함은 16S rRNA를 뜻한다. 조직 내에 존재하는 미생물이 물론 살아있는 것은 아니다. 그리고 장 이외의 원천에서 유입된 것도 적지 않다고 한다. 지금까지의 마이크로바이옴 연구의 방향을 근본적으로 뒤흔드는 이야기가 될 수도 있겠다.
서론이 지나치게 길었다. 사실 오늘 블로그 편집창을 연 것은 abricate라는 생소한 프로그램에 관해 알아보게 된 경위를 쓰려는 것이었다. TORMES 메인 스크립트를 좀 고쳐서 쓸 일이 생겼다. Python이 아니라 shell script라서 다행이었다. abricate라는 명령어가 항생제 내성 및 병원성 인자 유전자를 탐색하는 명령행에서 계속 나타나길래 도대체 이것이 뭔가 싶어서 구글에서 검색을 해 보았다. 여러 균주 샘플에서 나온 결과 파일을 한데 합치는 데에도 같은 프로그램이 쓰인다는 것이 흥미로웠다.
ABRicate
Mass screening of contigs for antimicrobial resistance or virulence genes. It comes bundled with multiple databases: NCBI, CARD, ARG-ANNOT, Resfinder, MEGARES, EcOH, PlasmidFinder, Ecoli_VF and VFDB.
ABRicate는 호주 멜버른 대학교의 Torsten Seemann의 작품이었다. Prokka, snippy 등 미생물 유전체를 다루는 사람이라면 밥 먹듯이 쓰는 프로그램의 개발자 아닌가. GitHub 사이트에서도 밝혔듯이 ABRicate의 첫 세글자는 Anti-Biotic Resistance에서 유래했다고 한다. 내가 특히 흥미를 느꼈던 것은 여러 샘플의 결과 리포트를 병합하여 매트릭스를 만들어주는 기능이 포함되어 있는 점이다('Combining reports across samples'). 만약 이를 셸 스크립트나 R로 하려면 얼마나 성가시겠는가? 커스텀 DB를 만들어 쓸 수도 있다는 점도 이 프로그램의 유용성을 잘 보여준다.
Seemann의 블로그 The Genome Factory를 종종 방문하여 유용한 정보가 있는지 알아봐야 되겠다. 흠, 올린 글은 그렇게 많지 않다. 하지만 그는 GitHub를 통해서 더욱 왕성한 활동을 하고 있으니 내가 도움을 받을 일은 앞으로도 많을 것이다.
댓글 없음:
댓글 쓰기