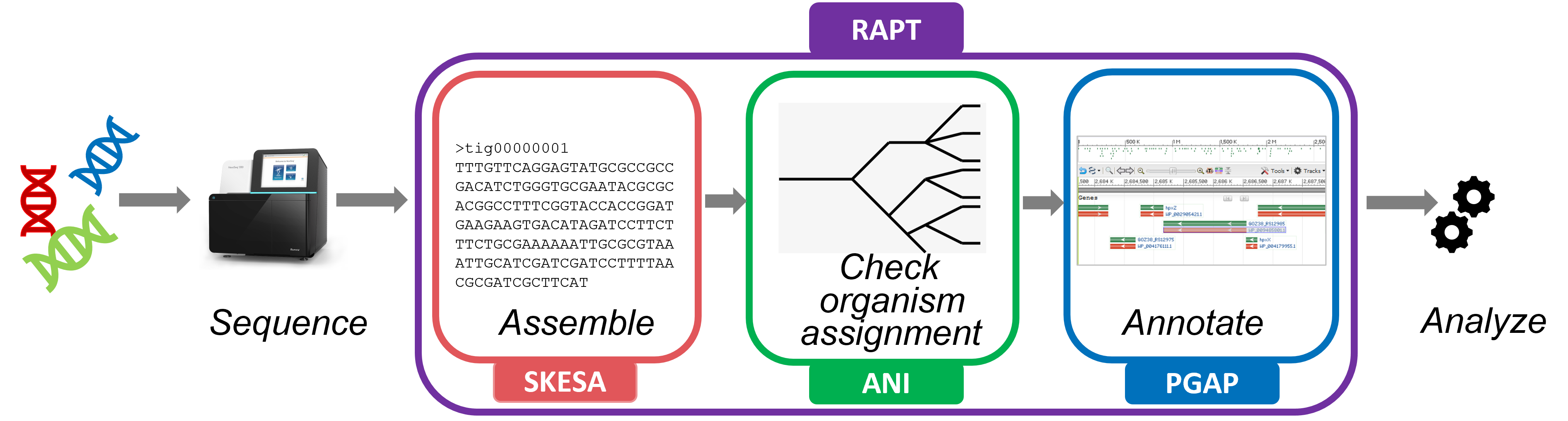
2021년 5월에 공개된 RefSeq release 206에서 뭐 새로운 것이 없는지 release notes를 읽다가 드디어 일루미나 read를 이용한 유전체 조립 도구를 서비스한다는 소식을 접했다. 이름은 RAPT. SRA accession을 입력해도 되고, 사용자가 직접 fastq/fasta 파일을 업로드해도 된다. 로그인이 필요한 서비스이다.
https://www.ncbi.nlm.nih.gov/rapt
 |
| RAPT Pipeline |
Benchmarking tools for de novo microbial assembly
오스트레일리아의 재주꾼 Torsten Seemann(prokka, snippy, abricate 등의 개발자)은 일루미나 read를 이용한 미생물 조립 도구인 Shovill을 내놓은바 있다. 엄밀이 말하면 이것은 SPAdes를 간편하게 쓰기 위한 wrapper에 해당하는데(metagenome assembly는 불가), 원한다면 SKESA나 Velvet 또는 Megahit 등 다른 assembler를 쓸 수도 있다.
NCBI의 서비스는 아주 발빠르지도 않고 화려하지도 않다. 그러나 한번 서비스를 개시하면, 매우 신뢰할 수가 있다. 또한 NCBI의 가장 큰 힘은 데이터의 보물창고라는 점 아니겠는가? 한국인이 개발한 genomics/bioinformatics 분야의 killer app이 있었던가? 정말 있으면 좋겠다. 아니, 한국의 데이터 저장고에만 있는 유용하고도 유일한 자료 같은 것이 있으면 더욱 좋겠다...
댓글 없음:
댓글 쓰기