Anvi'o는 각종 오믹스 정보를 분석하고 시각화하는 도구이다. 주로 다음과 같은 스타일로 분석 자료를 표현한다. 한 입을 베어먹은 도너츠와 같은 형태로 각종 데이터를 시각화한다.
2 년쯤 전에 메타게놈 분석 도구인 metAMOS에 강렬한 인상을 받고 이를 활용해 보고자 노력한 일이 있었다. 그러나 python 및 부수적으로 필요한 프로그램의 설치가 너무나 까다로와서 불완전한 상태로 설치를 마무리한 뒤 몇 번의 테스트를 하는 것으로 만족하고 말았다. 특히 shotgun assembly 기반의 metagenome data가 없으면 이를 활용하기도 어려웠다. 개발자 사이트를 가끔 가 보아도 별다른 업데이트에 대한 소식도 보이지 않았다.
그러던 중에 Anvi'o라는 독특한 프로그램을 발견하게 되었다. 이것도 기본적으로는 shotgun sequencing 기반의 metagenomics에 어울리는 도구이다. 단, co-assembly와 mapping은 사용자가 알아서 해야 된다. 그러나 pan-genome 분석과 phylogenomic 분석을 이 환경 안에서 할 수 있을뿐만 아니라 분석 결과를 웹브라우저에서 interactive하게 볼 수 있다.
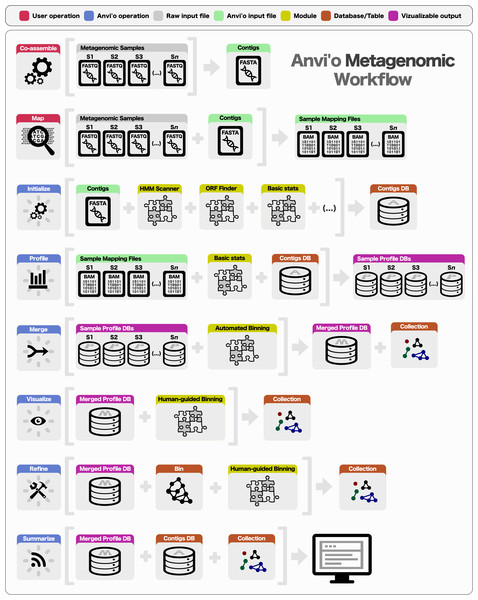 |
| Anvi'o의 metagenomic overflow. 빨강색 데이터는 유저가 준비해야 한다. Contig와 read mapping 파일이 이에 해당한다. 출처: PeerJ |
Anvi'o을 이제 실제로 다루어 봐야 되겠다고 다짐을 한 것은 오늘 아침이다. Bioconda를 이용하여 설치를 끝내고, 테스트 스크립트를 몇 개 돌려서 이상이 없음을 확인한 다음 최근에 논문으로 투고했던 Lactobacillus rhamnosus 111종 유전체 서열의 pangenomics analysis를 시작하였다. 꼭 필요한 과정은 아니지만 지금은 Anvi'o 환경 내에서 COG annotation을 실행하고 있다.
설치가 아주 쉽게 일사천리로 진행된 것은 아니다. cherrypy 실행과 관련한 에러, 그리고 CentOS 6.9에 크롬 대신 크로미움을 설치해야 하는 문제가 있었지만 metAMOS와 비교하면 아주 사소한 것들이었다. 무엇보다 고마운 것은 Anvi'o project page에 풍성하게 준비된 튜토리얼 자료들이었다. 몇 가지 핵심적인 기본 개념만 머리에 '탑재'하면 어렵지 않게 원하는 목적의 분석을 할 수 있는 것이다. 이에 대해서는 내 위키 페이지에 별도의 정보를 정리해 나가는 중이다.
 |
| Anvi'o 설치 후 테스트 스크립트 실행 화면. |
Phylogenomics 튜토리얼을 읽다가 내가 그동안 모르던 phylogenomic market set을 발견하였다. 이는 Campbell 등이 출간한 2011년 PNAS 논문 "Activity of abundant and rare bacteria in a coastal ocean(링크)"에서 소개한 것이다. 그동안 phylosift marker만 편식하던 나에게 매우 유용한 자양분이 될 것이다.
2018년 7월 6일에 추가한 글
어제의 테스트에서 anvi'o의 동작이 약간 이상하다고 생각했는데 그 원인은 python 2.7을 기반으로 anvi'o를 설치한 때문이었다. 이러한 중요한 정보는 설치 설명서 맨 위에 있었어야 하는 것 아닌가? Homebrew와 conda 등을 이용한 설치 설명에 이어서 '힘들게 설치하는 방법 - Installation (with varying levels of pain)' 파트에 있었던 다음의 경고문을 뒤늦게 발견했다. 그만큼 python 환경도 이제 3.x 버전이 대세임을 알아야 되겠다.
이미 설치가 되어있었던 python 3.5 기반 environment에 다시 설치를 하고 HMMER version을 3.1b2로 맞추니 모든 것이 정상적으로 돌아가기 시작하였다.

댓글 없음:
댓글 쓰기