여름에 우리나라에서 흔히 먹는 과채에서 유래한 RNA-seq data를 CLC Genomics Workbench에서 분석하려다가 아주 애를 먹었다. PASA/EVM에서 유래한 gene annotation file(GFF3)을 CLC에서 그대로 임포트하여 작업할 수 있는 상황이 아니었기 때문이다. GTF/GFF 파일을 처리하려면 먼저 sequence FASTA file을 임포트한 뒤
Annotate with GFF/GTF/GVF라는 플러그인을 사용하여 이를 FASTA에 덧입혀야 한다. 그러고나서
Convert to Tracks 명령을 사용하여 Sequence/CDS/Gene/mRNA track을 추출해야 한다. 여기까지 했는데 mRNA track이 생기질 않는 것이었다. 이 track은 annotation file의 mRNA 혹은 transcript feature line을 파싱하여 만들어지는 것이 아니라, 여러 exon feature가 같은 mRNA를 parent로 갖거나 동일한 transcript_id를 가질 경우 만들어지는 것이다. 주어진 GFF3 파일을 열어보면 exon feature가 갖는 parent는 mRNA가 아니라 "transcript"를 parent로 가리키고 있었다. 따라서 gene 바로 다음에 한 줄로 짜여진 transcript feature의 type을 mRNA로 바꾸니 제대로 읽혀지는 것을 확인하였다.
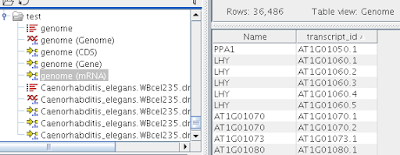
그 다음의 문제는 만들어진 mRNA feature를 구별하는 식별자(Name)를 결정하는 것이다. CLC에서는 다른 feature와 달리 transcript(mRNA)의 경우 부모 gene feature의 식별자를 기본으로 하여 일련번호를 붙인 것을 Name으로 재정의한다. Annotation file에서 몇가지 qualifier를 테스트해 보았으나, transcript_id를 도저히 name 컬럼으로 들어가게 할 수가 없는 것이었다. 다음 그림을 보라. 이것은 iGenomes ftp site에서 받은
Arabidopsis thaliana(TAIR10 기준)의 유전체 염기서열 파일 genome.fa에 유전자 annotation 파일인 genes.gtf를 덮어씌운 것이다. mRNA track에서 의 개별적인 transcript를 구별하는 식별자 Name은 gene symbol에서 온 것이다. LHY gene 위치에서는 총 5개의 transcript가 생성된다.
RNA-seq analysis 후에는 각 transcript_id를 기준으로 하여 expression value가 정리되면 정말 좋겠으나, 실제로는 LHY_1, LHY_2, LHY_3, LHY_4 그리고 LHY_5라는 새로운 체계의 식별자를 기준으로 한다. 밑줄 뒤에 붙은 번호는 transcript_id(AT1G01050.1) 뒤의 숫자와 아무런 관계가 없다.
그러므로 RNA-seq analysis를 마친 뒤에는 name이 아니라 transcript_id를 기준으로 expression value를 참고하면 된다. 이번에 작업한 GFF3 파일에는 transcript_id라는 qualifier가 아예 존재하지 않았다. transcript feature의 attribute 항목에 정의된 ID qualifier의 값을 그대로 이용하여 같은 feature line에 transcript_id를 추가하여 다시 실행을 하니 모두 정상적으로 작동을 하였다.
CLC Genomics Workbench를 쓴다고 해서 얄팍한 지식과 마우스 클릭만으로 손쉬운 분석을 할 수 있을 것이라고는 생각하지 말아야 되겠다.

댓글 없음:
댓글 쓰기