내 위키 사이트에 별도로 작성해 두었던 ANI 매트릭스 자료 분석법 문서를 수정하여 ComplexHeatmap의 Heatmap() 함수를 다루는 방법을 추가하였다. 앞으로 특별한 일이 없다면 gplots의 heatmap.2() 함수를 쓸 일은 없을 것 같다.
아직까지는 Heatmap() 함수 기능의 아주 일부분을 체험해 본 것에 불과하다.
 |
Heatmap의 색은 데이터 수치에 따라 파랑-하양-빨강으로 분포하게 되는데, 이를 하양-빨강의 단색 계열의 명암으로 나타낼 방법은 없을까? ComplexHeatmap Complete Reference의 7번 Legends 섹션을 계속 들여다 보았지만, 새로 추가하는 legend는 자유롭게 수정이 가능하지만 heatmap에서 자체적으로 생성되는 legend의 색을 바꾸는 방법이 보이지를 않았다.
이걸 사용자 입맛대로 바꾸지 못한다는 것은 말이 되지 않는다. 어디에 있을까... 아, 섹션 4의 A list of heatmaps를 둘러보니 하나의 플롯에 그려진 heatmap 두 개의 색상 체계가 다른 것을 발견하였다.
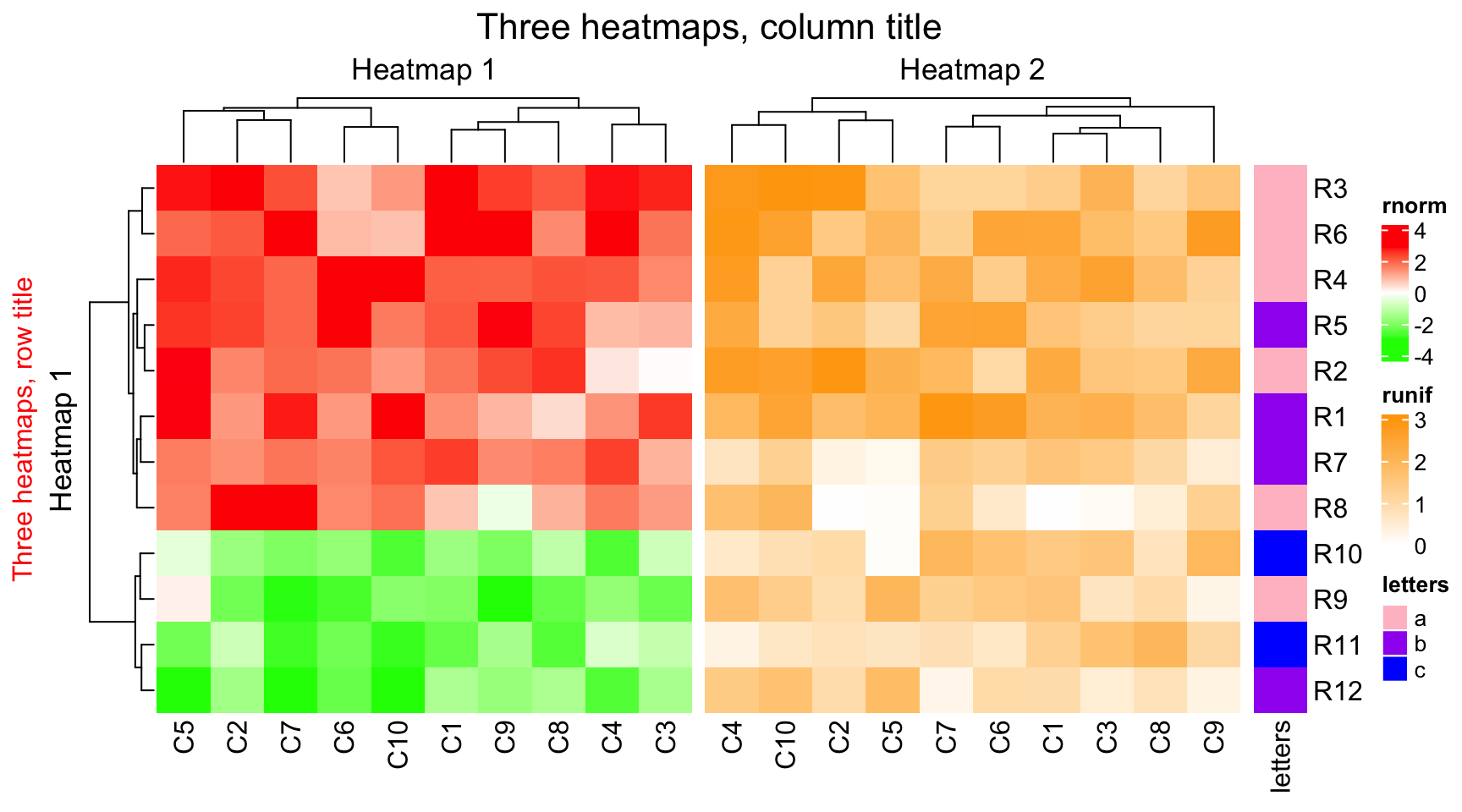 |
| 그림 출처 링크 |
가운데 heatmap을 그린 명령어를 확인해 보았다.
col_rnorm = colorRamp2(c(-3, 0, 3), c("green", "white", "red")) col_runif = colorRamp2(c(0, 3), c("white", "orange")) col_letters = c("a" = "pink", "b" = "purple", "c" = "blue") ht1 = Heatmap(mat1, name = "rnorm", col = col_rnorm, row_title = "Heatmap 1", column_title = "Heatmap 1") ht2 = Heatmap(mat2, name = "runif", col = col_runif, row_title = "Heatmap 2", column_title = "Heatmap 2") ht3 = Heatmap(le, name = "letters", col = col_letters) ht_list = ht1 + ht2 + ht3
두번째 줄이 유력한 용의자이다. colorRamp2() 함수는 circlize 패키지에서 제공하는데, 이는 ComplexHeatmap의 개발자인 "Bioinformagician" Zuguang Gu의 작품이기도 하다. 추앙하고 싶은 생명정보학자 리스트에 그의 이름을 올리겠다.
자, 그러면 흉내내기를 시작한다.
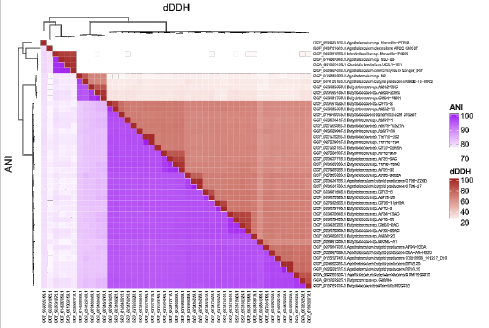
col_runif = colorRamp2(c(0.85, 1), c("white", "orange")) Heatmap(as.matrix(ani), name = "ANI", row_labels = key.2[rownames(ani)], row_names_gp = gpar(fontsize = 5), column_names_gp = gpar(fontsize = 5), col = col_runif)
| 화면 캡쳐하다가 column dendrogram이 잘렸다. |
다음 과제물 해결을 위한 힌트는 여기에서 얻도록 한다. 오늘은 힘들어서 더 이상은 못 하겠다.
Changing the default background color
2023년 2월 16일 업데이트
두 heatmap의 절반씩을 합쳐서 하나로 만들면 소원이 없겠네...
투명 이미지를 활용? 고민해 보자. 노동집약적인 방법을 동원한 결과는 다음과 같다.
포토샵을 잘 다루는 아들녀석에게 마무리를 위한 하청(?)을 주려다가 R 내에서 어떻게든 해 보기로 했다. 색상을 잘 선택하면 성격이 다른 두 자료를 하나의 매트릭스로 합쳐서 heatmap을 그려도 구별이 잘 되게 할 수 있을 것만 같다.

댓글 없음:
댓글 쓰기